LabSolutions Insight Biologics - Fonctionnalités
Logiciel de caractérisation des séquences d'oligonucléotides
Représentation de la formule structurelle
Dans la fenêtre de paramétrage des séquences oligonucléotidiques, la formule développée de la séquence saisie est affichée en temps réel, permettant une vérification rapide et simple des informations. En outre, les bases nucléiques utilisées dans la séquence, ainsi que les modifications de l'agent de liaison de base et du ribose peuvent être ajoutées et modifiées.
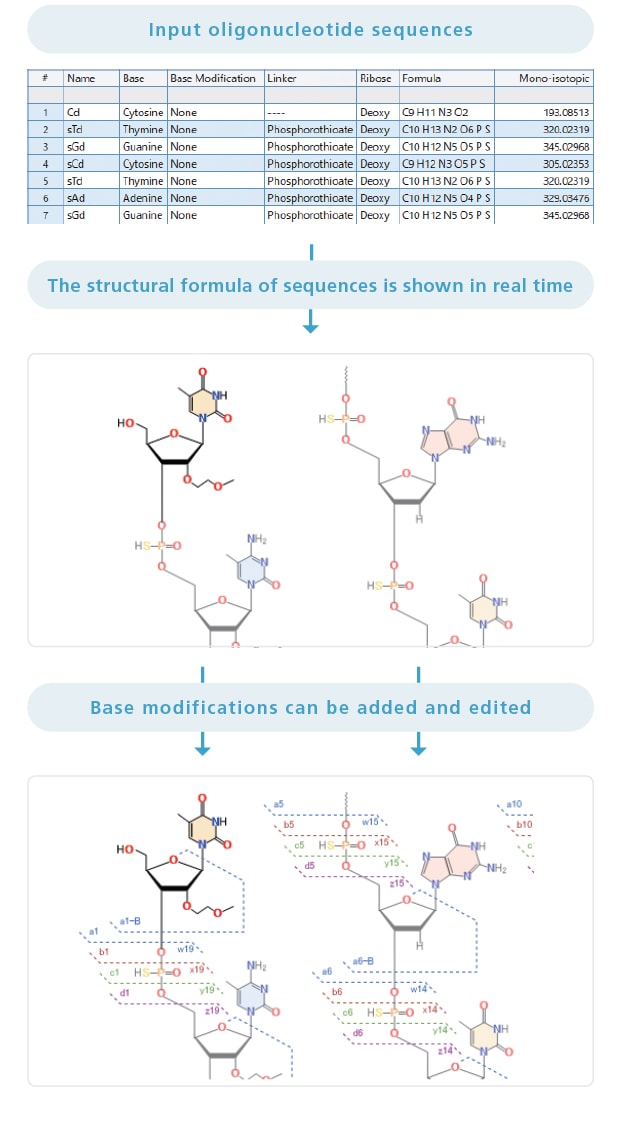
Affichage du chromatogramme des composants
Les pics d'impuretés sont affichés sous forme de chromatogramme de composants. Les chromatogrammes UV et MS peuvent être vérifiés simultanément.
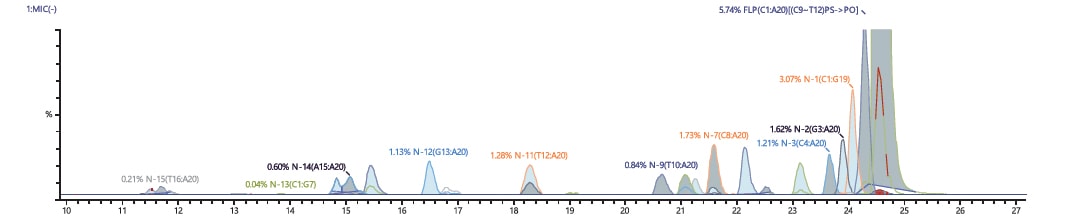
Affichage de la couverture des fragments
Le logiciel comprend un affichage de couverture qui indique les attributions spectrales des fragments. L’affichage de la couverture change pour correspondre aux éléments à vérifier. Des rapports peuvent également être générés.

Une expérience utilisateur supérieure
-
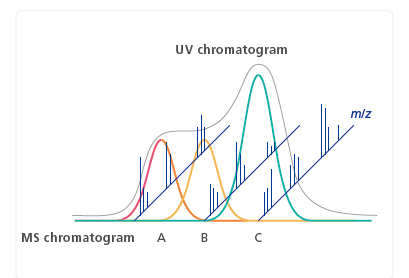
Calculs de pureté basés sur LC
L'utilisation simultanée des informations sur les pics du spectre MS avec les pics du chromatogramme LC intégré permet des calculs de pureté même pour les composants insuffisamment séparés dans le chromatogramme LC.
-
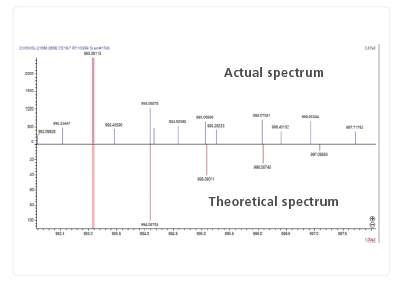
Fonction d'affichage des fragments
Le logiciel comprend un affichage de couverture, qui indique les affectations spectrales des fragments. L’affichage de la couverture change pour correspondre aux éléments à vérifier. Des rapports peuvent également être générés.
-
Optimisation des résultats d'identification
Le même composant est parfois détecté avec plusieurs temps de rétention, en fonction du pic d'intégration. La signification des résultats d'analyse problématiques est clarifiée par une optimisation facultative des résultats d'identification. De plus, la configuration de n’importe quel composant peut être déterminée à partir de tous les ions détectés, il n’est donc pas nécessaire d’examiner les tableaux générés pour chaque valence.
-
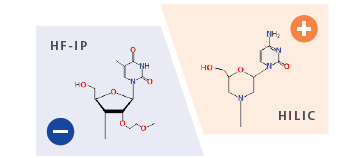
Fonctions d'analyse pour chaque polarité d'ionisation
La polarité d'ionisation permet d'analyser les données non seulement du spectre d'ionisation par polarité négative à l'aide de réactifs à paires d'ions, qui est la méthode traditionnelle, mais également du spectre d'ionisation de polarité positive via HILIC. Le logiciel peut analyser l’acide nucléique morpholino, difficile à ioniser négativement.
-
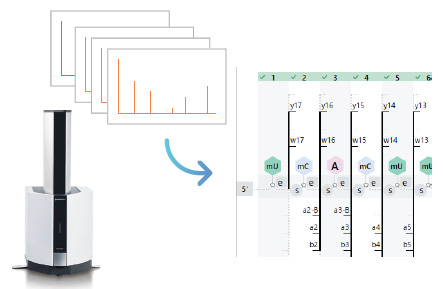
Analyse efficace des spectres MALDI
Avec MALDI-MS, l'identification des composants et l'estimation de la séquence sont effectuées en sélectionnant un spectre d'identification à l'aide de données exportées au format mzML et jusqu'à trois spectres de fragments. L'algorithme d'analyse des données mis en œuvre est adapté aux instruments caractérisés par un spectre univalent. Même si un spectre MALDI est utilisé, les procédures de confirmation des résultats d'identification sont les mêmes que pour les autres instruments. Le logiciel a été conçu pour être transparent, il n'est donc pas nécessaire de prendre en compte les coûts de formation liés à l'exploitation.
-
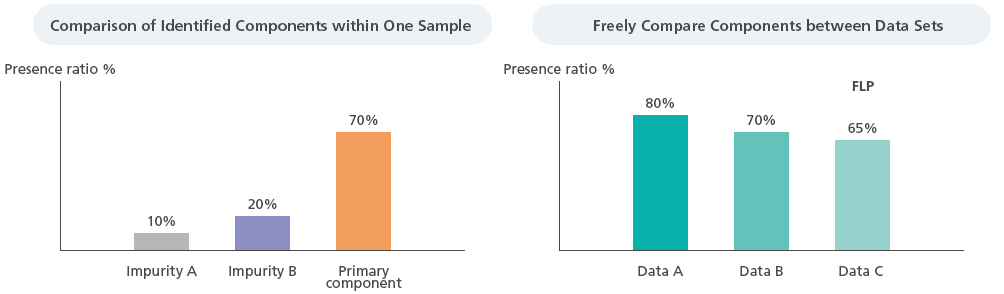
Fonctions de comparaison de composants
Les résultats d'identification au sein d'un échantillon et pour chaque ligne de lot peuvent facilement être comparés.
Prise en charge de l'intégrité des données
LabSolutions Insight Biologics contribue à accroître l'efficacité du développement tout en gérant les données conformément aux directives de la FDA américaine 21 CFR Part 11, de l'édition japonaise des directives ER/ES et de l'annexe 11 des BPF de l'UE.


