Analyse de la séquence d'acides aminés N-terminaux par MALDI-TOF MS
Analyse de la séquence d'acides aminés N-terminaux
Analyse de la séquence d'acides aminés N-terminaux par MALDI-TOF MS
Les fragments N-terminaux peuvent être isolés même à partir de protéines dont les N-terminaux sont bloqués. Les fragments N-terminaux de protéines avec des N-terminaux non bloqués peuvent être récupérés sous forme de dérivés d'acide sulfonique, ce qui permet un séquençage de novo par MALDI-MS/MS. Les fragments N-terminaux peuvent être isolés séparément même si plusieurs protéines coexistent dans un seul échantillon SDS-PAGE (bande).
Kit de séquençage de masse ORFinder-NB (kit de séquençage des protéines N-Terminal)
- Isole les fragments N-terminaux des digestions par la trypsine des protéines à l'aide de résines DITC, que les N-terminaux des protéines soient bloqués ou non.
- Récupère les fragments N-terminaux de protéines avec des N-terminaux non bloqués en tant que dérivés de l'acide sulfonique. En conséquence, ce sont principalement les ions de séquence y qui sont détectés dans les spectres MS/MS, permettant une analyse de séquence de novo .
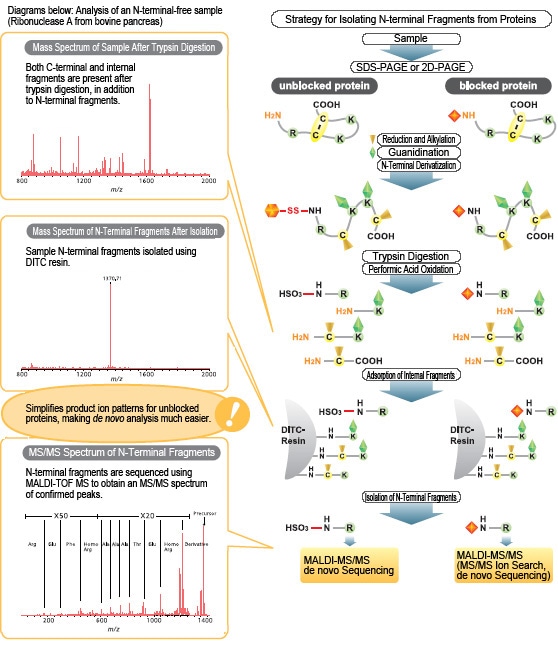
Kit de séquençage de masse ORFinder-NB (kit de séquençage des protéines N-Terminal)

Permet l'isolement de fragments N-terminaux, même à partir de protéines dont les N-terminaux sont bloqués. Les fragments N-terminaux de protéines avec des N-terminaux non bloqués peuvent être récupérés sous forme de dérivés d'acide sulfonique, ce qui permet un séquençage de novo par MALDI-MS/MS. Les fragments N-terminaux peuvent être isolés séparément même si plusieurs protéines coexistent dans un seul échantillon SDS-PAGE (bande).
Spectromètre de masse à temps de vol à désorption/ionisation laser assisté par matrice MALDI-TOF MS

MALDI-TOF MS AXIMA Performance est un MALDI-TOF MS de type TOF-TOF doté d'un CID à haute énergie.
Permet d'obtenir une sensibilité de mesure MS/MS encore plus élevée, offrant une grande fiabilité pour l'analyse du protéome. La MS automatisée (ou MS/MS) sur plusieurs échantillons combinée aux recherches dans la base de données Mascot permet l'identification rapide des marqueurs protéiques candidats.


