Analyse lipidique d'un cerveau de souris par un logiciel d'analyse statistique
L’élément le plus important de l’imagerie MS est l’efficacité avec laquelle elle peut analyser les énormes quantités de données acquises par acquisition de données.
Imaging MS Solution Analysis est un logiciel d'analyse d'images dédié à iMScope qui offre une détection facile des pics, une analyse en composantes principales (PCA), une analyse de cluster hiérarchique (HCA) et une analyse de région d'intérêt (ROI). La détection des pics et l'ACP ont été effectuées sur des données d'imagerie MS d'une tranche de cerveau de souris afin d'obtenir des groupements lipidiques présentant les trois types différents de caractéristiques de distribution correspondant aux structures cérébrales. Il a été démontré que la superposition d'une micrographie sur ces distributions lipidiques permet de visualiser les distributions localisées.
* Zone de vente : Toutes zones hors Amérique du Nord
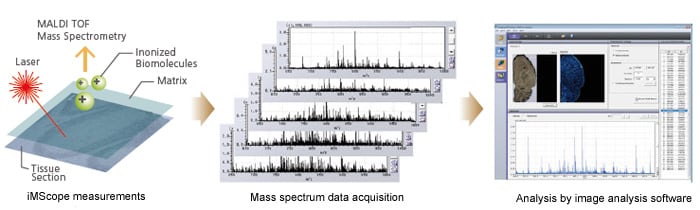
Imagerie MS et analyse de données
L'imagerie MS consiste à utiliser un spectromètre de masse pour capturer les matériaux ionisés générés par une irradiation continue par de fins tirs laser à travers l'espace bidimensionnel d'une tranche de tissu sous forme de spectre de masse. Des images planaires sont créées à partir du m/z, des informations de position et des intensités de signal dans les données obtenues.
Le microscope de masse d'imagerie iMScope peut acquérir jusqu'à 250 x 250 spectres de masse de pixels à partir de chaque acquisition de données à une résolution offrant un pas minimum de 5 μm et permettant l'acquisition de données à partir de régions extrêmement petites. L'iMScope est fourni avec le logiciel d'analyse d'images dédié Imaging MS Solution Analysis pour une analyse précise des données.
Logiciel d'analyse d'images "Imaging MS Solution Analysis"
Le logiciel d'analyse d'image « Imaging MS Solution Analysis » utilisé avec le microscope de masse d'imagerie iMScope offre la détection des pics, l'analyse en composantes principales (ACP), l'analyse de grappes hiérarchiques (HCA) et l'analyse des régions d'intérêt (ROI). Il réalise cette gamme diversifiée d’analyses sans avoir besoin d’une configuration fastidieuse des paramètres des méthodes analytiques.
L'analyse en composantes principales (ACP) extrait la matrice des pics du spectre de masse et recherche les composants principaux comme modèles caractéristiques dans les images. Il détecte les valeurs avec une distribution spécifique, puis recherche d'autres valeurs avec une distribution similaire.
L'analyse de cluster hiérarchique (HCA) compare les images m/z pour analyser des images m/z similaires sous forme de clusters (groupes). La distance entre les clusters (facteur de mésappariement) est utilisée pour évaluer la similarité de chaque cluster. La similarité peut être confirmée en affichant l’arborescence et les informations sur les facteurs de non-concordance.
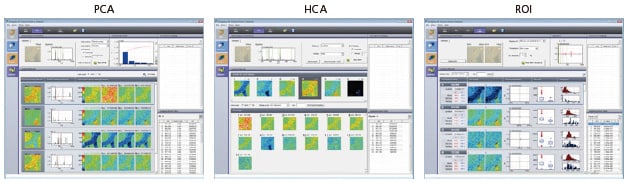
Fig.1 Logiciel d'analyse de données
L'analyse de la région d'intérêt (ROI) est effectuée pour tester les différences d'intensité entre plusieurs ROI désignées ou une ROI intérieure et extérieure dans un seul lot de données d'imagerie. La valeur p indique la probabilité qu’aucune différence d’intensité n’existe, par rapport à l’hypothèse nulle selon laquelle aucune différence d’intensité ionique n’existe. Plus la valeur p est petite, plus la probabilité qu’une différence d’intensité existe est élevée ; c'est-à-dire si chaque région spécifiée comme ROI contient des molécules avec des valeurs m/z qui ont une distribution caractéristique. Par exemple, effectuer une analyse de la ROI avec une ROI distincte spécifiée pour les régions cancéreuses et non cancéreuses au sein du même tissu peut détecter des molécules spécifiques à la région cancéreuse.
Imagerie MS d'une tranche de cerveau de souris
Conditions d'analyse
Tableau 1 : Produits pharmaceutiques cibles et performances de la méthode
| Échantillon | Tranche de cerveau de souris (environ 10 μm d'épaisseur) |
| Lame de verre | Verre ITO (Sigma Aldrich #578274) |
| Instrument de mesure | iMScope |
| Matrice | DHB (pulvérisation) |
| Nombre de pixels | 110x213 |
| Résolution spatiale | 50 μm |
| Plage de mesure (m/z) | 650.000 à 1.000.000 |
| Entrain de mesurer le temps | 7 minutes |
| Cueillette de pointe | Automatique (100 pics) Hors certains pics matriciels |
| Analyse ACP | Automatique (réglage par défaut) |
Sur la pose d'une micrographie
La figure 4 montre la micrographie superposée aux espèces moléculaires localisées caractéristiques dans chaque région identifiées par analyse PCA.
Bien que cette analyse ait été réalisée à une résolution spatiale de 50 μm, il est évident qu’une bonne correspondance a été obtenue entre les distributions moléculaires et la micrographie, même dans les régions à structure extrêmement fine, telles que le corps calleux et l’hippocampe. Le logiciel Imaging MS Solution Analysis offre des fonctions d'affichage pour la superposition d'images optiques et d'images m/z et la superposition de plusieurs images m/z. Il affiche facilement des images de superposition précises, sans avoir besoin d'ajustements de position détaillés. Des images de superposition similaires sont également disponibles pour les données d’analyse MS-MS.
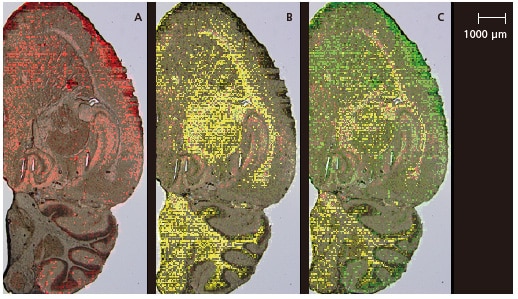
Fig. 4 Images optiques superposées

Fig. 2 Imagerie MS/MS de la rétine de rat, montrant l'espace (distribution de la chloroquine m/z 320,190 247,095)
Microscope de masse à imagerie iMScope TRIO

L'imagerie par spectrométrie de masse est une nouvelle technologie révolutionnaire.
L'instrument est une combinaison d'un microscope optique qui permet l'observation d'images morphologiques à haute résolution, avec un spectromètre de masse qui identifie et visualise la distribution de molécules spécifiques.


