Analyse du séquençage des acides aminés N-terminaux par MALDI-TOF MS/Séquenceur de protéines
La spectrométrie de masse est devenue un outil indispensable pour les chercheurs cherchant à séquencer des peptides. Bien qu'efficace dans de nombreux cas, le séquençage par In Source Decay (ISD) se heurte à quelques défis liés à sa capacité à fournir des informations de séquence fiables, notamment les acides aminés isobares, la dépendance à la base de données et les interférences de faible poids moléculaire.
Le séquençage Edman traditionnel évite la dépendance de masse et l’utilisation de bases de données en analysant chaque acide aminé de l’extrémité N-terminale un par un dans l’ordre. Malheureusement, Edman a ses propres limites pour fournir une couverture de séquences élevée.
Cette note technique étudie les avantages de la combinaison des informations de masse intacte et de séquençage du MALDI-8020 (ISD) avec la séquence N-terminale obtenue à partir du système de gradient PPSQ-50A (Edman). L’utilisation des informations combinées permet aux enquêteurs d’obtenir une image plus complète de leurs protéines et peptides d’intérêt.
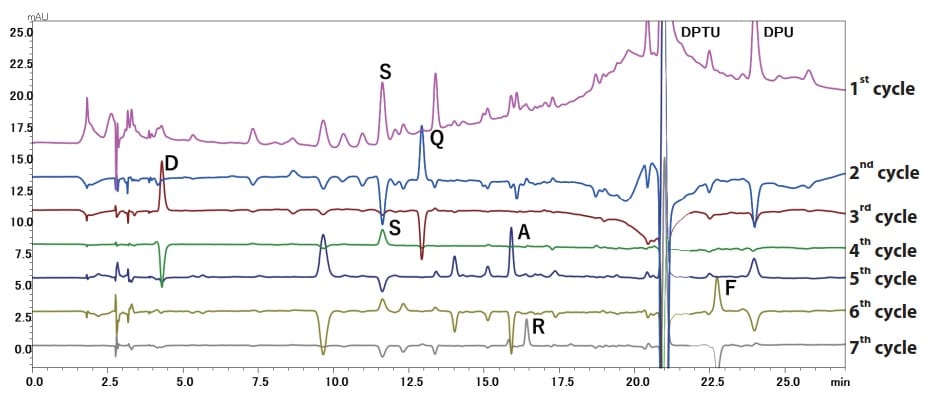
Fig. 1 Subtracted Chromatograms from First Seven Cycles Using the PPSQ-50A Gradient System
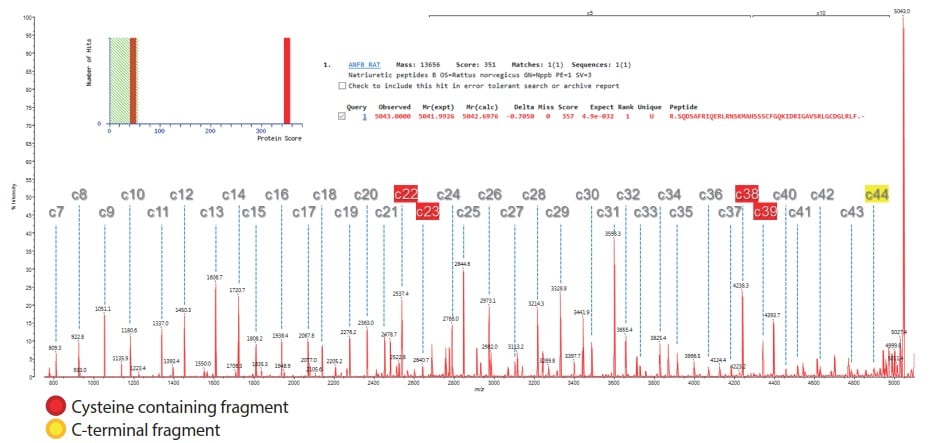
Fig. 2 Spectre ISD du BNP utilisant MALDI-8020 avec la matrice DAN. Identification de la MASCOTTE affichée en médaillon
■ Actualités relatives aux applications ■
Séquenceur de protéines

Caractéristiques :
Le PPSQ-51A/53A perpétue la tradition consistant à fournir aux chercheurs un séquençage fiable et sensible des protéines N-terminaux grâce à la dégradation Edman automatisée. Qu'il s'agisse de passer d'un autre séquenceur ou d'adopter la technique pour la première fois, le logiciel intuitif et le matériel Shimadzu robuste faciliteront le séquençage des protéines dans votre laboratoire.



